김기동 분당서울대병원 산부인과 교수. [사진=분당서울대병원]
암 조직이 끈적한 점액질로 이뤄진 ‘점액성 종양’의 경우 표준 치료 과정을 적용하기 매우 힘들다. 일반적인 종양과는 달리 다른 장기에서 전이되더라도 세포 모양이 유사하고, 발현 물질의 차이가 크게 없어 원발 부위를 특정하기 어렵기 때문이다.
김기동 분당서울대병원 산부인과 교수팀은 암세포가 기원한 장기에 따라 RNA(리보핵산)의 발현 패턴이 다르다는 점에 착안, ‘전사체 분석’이 정확한 검사법으로 활용될 수 있는지 규명하는 연구를 수행했다. 전사체는 한 세포 내에 존재하는 모든 RNA 분자의 총합이다.
연구팀은 1960개의 암 검체의 전사체 데이터를 바탕으로 자궁경부암, 자궁내막암(자궁체부암), 난소암, 자궁암육종, 췌장암, 위암, 대장암 등 7개 원발 암에 따라 각기 다르게 발현하는 RNA 패턴을 기계학습 시켜, 점액성 종양의 원발부위를 찾아내는 알고리즘을 개발하는 데 성공했다. 정확도는 약 85.7% 수준이다.
이번 연구 결과는 세이지(SAGE) 출판사에서 발행하는 국제학술지 캔서 인포매틱스(Cancer Informatics)에 게재됐다.









![[포토] 석촌호수에 나타난 포켓몬 라프라스와 피카츄](https://image.ajunews.com/content/image/2024/04/26/20240426155410280572_518_323.jpg)
![[포토] 악수하는 이재명 대표와 조국 대표](https://image.ajunews.com/content/image/2024/04/26/20240426003606867599_518_323.jpg)
![[포토] 최정, 한국 야구 역사 468호 쾅](https://image.ajunews.com/content/image/2024/04/24/20240424232935147850_518_323.jpg)
![[슬라이드 포토] 성수동이 들썩! 입생로랑 뷰티 팝업 방문한 스타들](https://image.ajunews.com/content/image/2024/04/24/20240424184737983118_518_323.jpg)
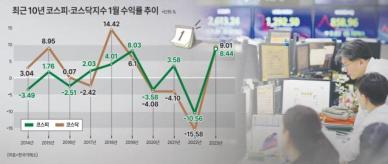
![[금투세 폐지 논란] 코리아 디스카운트 심화...韓증시 불확실성 높여](https://image.ajunews.com/content/image/2024/01/03/20240103150426112900_388_136.jpg)